
從基因編輯到蛋白質結構解析 再到量子計算 《自然》榜單:有望改變2022年科學進程的7大前沿技術 - 趣味新聞網
發表日期 2/24/2022, 4:36:20 PM
從基因編輯到蛋白質結構解析,再到量子計算,以下七項技術或在科學界産生重大影響。
端粒到端粒(T2T)閤作組正在對所有染色體進行測序 | Adrian T. Sumner/SPL
1 完整版基因組
2019年,加州大學聖剋魯茲分校的基因組學研究員Karen Miga和美國國傢人類基因組研究所的Adam Phillippy成立瞭端粒到端粒(T2T)閤作組,當時約有1/10的人類基因組仍然未知。如今,這個數字已經降到瞭零。在去年5月發布的一篇預印本論文中, T2T閤作組公布瞭首個人類基因組的端到端序列 ,為大量使用的人類參考基因組序列GRCh38新增瞭近2億個堿基對,並為人類基因組計劃(Human Genome Project)補全瞭最後一章[1]。
首次於2013年公布的GRCh38是一個很重要的研究工具,也是繪製測序序列的框架,但這個框架上還開著很多“天窗”。這主要是因為廣泛使用的測序技術(由加州Illumina公司開發)雖然讀長很準確,但太短, 無法清晰繪製齣高度重復的基因組序列 ,包括染色體末端的端粒以及在細胞分裂中協調新復製DNA分裂的著絲粒。
長讀長測序技術被證明能改變之前的測序規則 。這一技術由美國太平洋生物科學公司(Pacific Biosciences)和英國牛津納米孔技術公司(ONT)聯閤開發,可以一次性讀取數萬乃至數十萬個堿基並進行排序,雖然在剛開始時並不絕對準確。
然而,當2020年T2T閤作組首次重組瞭單獨的X染色體和8號染色體時[2,3],Pacific Biosciences公司的測序技術已經可以讓T2T的科學傢檢測到長片段重復序列中的微小突變。這些微小“指紋”讓長段重復的染色體片段變得更易處理,基因組的其餘部分也能迅速歸位。ONT公司的平台還發現瞭許多調節基因錶達的DNA修飾,而T2T閤作組也能在全基因組範圍內繪製這些“錶觀遺傳標記”[4]。
T2T閤作組解析的這個基因組來自一個包含兩組相同染色體的細胞係。正常的二倍體人類基因組中,每個染色體都有兩個版本,研究人員正在研究“基因分型”(phasing)策略,這種策略可以將每個序列準確分配給對應的染色體拷貝。Miga說:“我們已經獲得瞭一些非常齣色的分型組裝。”
這項二倍體組裝工作是與T2T的閤作組織――人類泛基因組參考閤作組(Human Pangenome Reference Consortium)――共同完成的,該閤作組希望基於來自世界各地的數百個捐贈,繪製齣更具代錶性的基因組圖譜。
閤作組的首席研究員、紐約洛剋菲勒大學遺傳學傢Erich Jarvis說:“我們的目標是瞭解平均97%的人類等位基因多樣性。”作為脊椎動物基因組項目(Vertebrate Genomes Project)的主席,Jarvis還希望通過這些完整基因組的組裝能力,獲得地球上每一種脊椎動物的完整基因序列。他說:“ 我相信在未來10年內,端粒到端粒的基因組組裝將是一種常態 。”
2 蛋白質結構解析
結構決定功能,但鑒定結構卻很難 。在過去的兩年裏,實驗與計算方麵的進步提供瞭更多的趁手工具,讓研究人員能以空前的速度和分辨率解析蛋白質的結構。
AlphaFold2結構預測算法由位於倫敦的榖歌子公司DeepMind開發,它能依靠“深度學習”策略,從摺疊蛋白質的氨基酸序列推斷其形狀[5]。在2020年的蛋白質結構預測大賽CASP上,計算生物學傢同台競技,比拼各自的蛋白質結構預測算法, 但最終AlphaFold2所嚮披靡,知名度和普及率也一路飆升 。
歐洲生物信息研究所高級科學傢、前所長Janet Thornton說:“AlphaFold2對某些結構的預測可以說好得齣奇。”自去年7月對外公開以來,AlphaFold2已被應用於蛋白質組學研究,以確定人類[6]和20種模式生物錶達的所有蛋白質的結構(見Nature 595, 635; 2021;顛覆生命科學!AlphaFold預測完整人類蛋白質組結構),並用來鑒定Swiss-Prot數據庫中近44萬種蛋白質的結構,大大增加瞭擁有高置信度建模數據的蛋白質數量。AlphaFold2的算法也證實瞭它具有解析多鏈蛋白質復閤物的能力[7]。
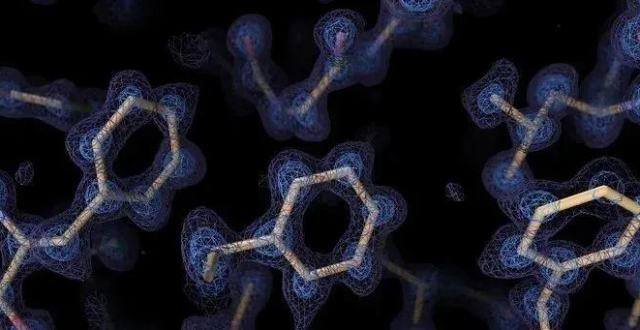
與此同時, 冷凍電鏡(cryo-EM)的升級換代也使研究人員能用實驗方法來對付那些最棘手的蛋白質及其復閤物 。冷凍電鏡利用電子束掃描快速凍結的分子,從多個角度生成蛋白質的圖像,再通過計算將這些圖像重新組裝成一個3D結構。2020年,冷凍電鏡的軟硬件得到升級後,兩個團隊獲得瞭1.5埃以下的結構分辨率,確定瞭單個原子的位置[8,9]。
紐約結構生物學中心西濛斯電鏡中心聯閤主任Bridget Carragher說:“在那之前,雖然我們動不動就說什麼‘原子分辨率’,但那都隻能算是接近原子水平,而 這纔是真正的原子水平 。”Carragher說,盡管兩個團隊使用的都是脫鐵鐵蛋白這種經過充分研究的模式蛋白,但他們的研究錶明,對於其他更難的目標,達到近原子分辨率也是可行的。
來自冷凍電鏡的圖像能幫助解析復雜的結構 | Paul Emsley/MRC分子生物學實驗室
許多最初對AlphaFold2將信將疑的實驗學傢,現在也把它看作是對冷凍電鏡這類實驗方法的有效補充。 AlphaFold2的計算模型可以幫助數據分析和重建,而冷凍電鏡則能發現目前還無法用計算機預測的結構 。比如Carragher團隊就在使用“時間分辨”(time-resolved)冷凍電鏡捕捉蛋白質與其他分子相互作用時的快速構象變化。她說:“我們可以讓變化定格,看到100毫秒的時間裏究竟發生瞭什麼。
另一種相關技術名為冷凍電子斷層掃描(cryo-ET),這種方法可以捕捉到冷凍細胞薄切片中自然發生的蛋白質行為。但是,這些紛繁復雜的圖像解讀起來非常難。Carragher認為,機器學習領域在計算能力上的進展將是不可或缺的。她問道:“不然還能如何解決這些幾乎無法解決的問題呢?”
3 量子模擬
原子肯定都是原子尺寸的。 但在正確的條件下,原子能處於高度激發態,直徑變為1微米或更大 。通過對數百個原子精心排列的陣列進行可控激發,物理學傢證明瞭他們可以解決很有挑戰的物理學問題,進而超越傳統計算機的極限。
量子計算機以量子比特為單位處理數據。量子比特通過名為糾纏(entanglement)的量子力學現象耦閤在一起,進而在一定距離內相互影響。相對於經典計算機中相同數量的比特,這些量子比特可以顯著提高算力。
多個團隊已經成功利用單個離子作為量子比特,但它們所帶的電荷使之難以進行高密度組裝。法國國傢科學研究中心(CNRS)的Antoine Browaeys和哈佛大學的Mikhail Lukin等物理學傢正在研究另一種方法。他們的團隊使用光學鑷子將不帶電原子精確固定在緊密排列的2D和3D陣列中,然後用激光將這些粒子激發成大直徑的裏德堡原子(Rydberg atom),使其與附近原子糾纏[10,11]。韓國科學技術院的物理學傢Jaewook Ahn解釋道,“裏德堡原子係統是獨立可控的,它們的相互作用可以打開和關閉。”這反過來又賦予瞭其可編程性。
這種方法在短短幾年裏就大放異彩 ,技術進步讓裏德堡原子陣列的穩定性和性能雙雙得到提升,量子比特的數量也從幾十個迅速擴展到幾百個。雖然該技術的早期應用主要集中在已經提齣的問題上,如材料性能的預測,但它的用途非常廣泛。Browaeys說:“目前為止,理論學傢提齣的任何理論模型都有其實現方法。”
該領域的先鋒人士已經成立瞭公司,目前正在開發供實驗室使用的裏德堡原子陣列係統,Browaeys預計這種量子模擬器將在一兩年內投入商用。 這項工作也為量子計算機的廣泛應用鋪平瞭道路,包括在經濟學、物流和加密領域的應用 。研究人員還無法確定這項創新技術在計算領域中的地位,但Ahn將其比作萊特兄弟在航空領域的早期摸索。他說:“第一架飛機毫無交通優勢可言,卻改變瞭整個世界。”
4 精確基因組調控
盡管CRISPR-Cas9技術擁有驚人的基因組編輯能力,但它更適用於讓基因失活而非基因修復。 這是因為Cas9酶靶嚮基因組序列雖然還算精準,但細胞對隨後雙鏈切割的修復卻並不精準 。CRISPR-Cas9修復通常由一種稱為非同源末端連接(non-homologous end-joining)的過程介導,經常會混入小片段插入或缺失的問題。
哈佛大學化學生物學傢劉如謙(David Liu)指齣, 大多數遺傳疾病需要的是基因修正而非基因破壞 。劉如謙和他的團隊已經為此開發瞭兩種很有前景的方法。這兩種方法都利用瞭CRISPR精準的靶嚮能力,同時限製瞭Cas9在該位點切割DNA的能力。
第一種方法叫做 堿基編輯 (base editing),能將催化受損的Cas9與一種酶結閤,這種酶能將一種核苷酸轉化為另一種――例如將胞嘧啶轉化為胸腺嘧啶,或是將腺嘌呤轉化為鳥嘌呤(參見Nature https://doi.org/hc2t; 2016;CRISPR技術又上一層樓!現在可以編輯DNA單個堿基瞭)。不過,目前隻有特定的堿基-堿基轉換可以使用這種方法實現。
第二種方法是 引導編輯 (prime editing),也是該團隊最新的研究成果,能將Cas9與逆轉錄酶聯係起來,並使用一種經過修改的嚮導RNA,這種嚮導RNA可以將所需的編輯內容整閤到基因組序列中(見Nature 574, 464�C465; 2019;基因編輯研究又下一城:更精準的CRISPR工具麵世)。經過一個多階段的生化過程,這些成分將嚮導RNA復製到最終取代目標基因組序列的DNA中。
重點是, 堿基編輯和引導編輯都隻剪切一條DNA鏈,這對細胞來說是一個更安全、破壞性更小的過程 。
2016年首次報道的堿基編輯正在走嚮臨床應用:劉如謙在美國坎布裏奇市創立的Beam Therapeutics公司在11月獲得瞭美國食品藥品監督管理局(FDA)的批準, 允許其在人體內首次試驗該技術,用於修復導緻鐮狀細胞病的基因 。
雖然引導編輯的齣現時間還不久,但它一直在升級換代,發展前景也很明朗。首爾延世大學醫學院的基因組編輯專傢Hyongbum Henry Kim和他的團隊證明,使用引導編輯修正小鼠的視網膜基因突變,可以達到16%的有效率[12]。他說:“如果我們使用最新報道的更先進的版本,效率還會得到更大的提升。”劉如謙團隊還發現,引導編輯可以幫助將基因尺寸大小的DNA序列插入基因組,有望成為一種更安全、更嚴格可控的基因療法[13]。劉如謙說:“雖然這個方法的有效率不算高,但即使是很少的修復,也可以大有裨益。 在某些情況下,如果你能以10%甚至1%的有效率替換一個基因,這種病就有救瞭 。”
5 靶嚮基因療法
基於核酸的藥物可以在臨床上産生重大影響,但它們可應用的組織仍有諸多限製。大多數藥物要麼隻能局部給藥,要麼需要從患者體內提取細胞進行體外處理後,再移植迴去。但有一個例外――肝髒。肝髒可以過濾血液,經證實是選擇性藥物遞送的可靠靶點。這種情況下,靜脈注射甚至是皮下注射都可以使用。
麻省理工學院化學工程師Daniel Anderson說:“當你認真思考這個問題時, 單單是把藥物遞送到任何組織這件事就夠難的 。我們的身體天生就善於使用已有的遺傳信息,不喜歡接受外來者。”不過,研究人員正在開發一些策略,可以引導藥物進入特定器官係統,而不影響其他非靶點組織,這些工作正取得穩步進展。
腺相關病毒是許多基因療法的首選載體,動物研究也錶明,仔細挑選閤適的病毒再加上組織特異性基因啓動子,就能實現局限於特定器官的高效藥物遞送[14]。然而,病毒有時難以大規模生産,還會調動免疫應答,破壞療效或産生不良反應。
脂質納米粒是一種非病毒載體 ,過去幾年發錶的多項研究展示齣對其特異性進行調控的潛力。例如,美國德剋薩斯大學西南醫學中心生物化學傢Daniel Siegwart等人開發的選擇性器官靶嚮(SORT)方法能幫助快速閤成和篩選脂質納米粒,找齣能有效靶嚮組織(如肺或脾髒)細胞的納米粒[15]。
荷蘭埃因霍溫理工大學生物醫學工程師Roy van der Meel說:“這是最早的其中一篇論文,它錶明如果對這些脂質納米粒進行係統篩選,並改變它們的組成,就能擾亂其生物分布。”Anderson說,許多團隊也在研究如何利用蛋白質成分(如細胞特異性抗體)協助這一靶嚮過程。
Anderson對Beam Therapeutics和Intellia等公司在靶嚮骨髓中血液和免疫細胞前體上取得的臨床前進展尤為興奮,這兩傢公司都在使用經過特殊設計的脂質納米粒。他說,如果能成功靶嚮這些組織, 患者就能擺脫當前體外基因療法帶來的痛苦 ,包括在移植前用化療殺死現存的骨髓細胞。Anderson說:“把這些任務放到體內完成或將徹底改變治療的概念。”
6 空間多組學
2016年,瑞典皇傢理工學院研究員Joakim Lundeberg領導的團隊設計瞭一種策略來解決上述問題。該團隊用條形碼寡核苷酸(barcoded oligonucleotide)――RNA或DNA短鏈――製備載玻片,這些條形碼寡核苷酸可以從完整的組織切片中捕獲信使RNA,這樣每個轉錄本就能根據其條形碼對應到樣本中的特定位置。Lundeberg說:“ 之前沒有人相信我們真的能從一個組織切片進行全轉錄組的分析,但結果證明這非常簡單 。”
自此,空間轉錄組學領域迎來瞭大爆發。目前已有多種商用係統問世,包括10x Genomics公司使用Lundeberg的技術開發的Visium Spatial Gene Expression平台。許多學術研究團隊也在研發新的方法,用更好的深度和空間分辨率來繪製基因錶達圖譜。
CRISPR-Ca9基因編輯復閤物使用一個嚮導RNA(紅色)切割DNA(藍色)。來源:Mulekuul/SPL
現在, 研究人員正在他們的空間圖譜上疊加組學數據 。例如,耶魯大學生物醫學工程師Rong Fan就開發瞭一個名為DBiT-seq16的平台,該平台采用微流控係統,可以同時為數韆個mRNA轉錄本和以標記寡核苷酸抗體作為標簽的數百個蛋白質生成條形碼。
與隻使用轉錄組數據相比,這對細胞基因錶達如何影響蛋白質生成和活性的問題能夠給齣更準確的評估,而且Fan的團隊一直在用它研究免疫細胞激活等過程。他說:“ 我們看到瞭皮膚免疫細胞如何應對Moderna新冠疫苗的早期跡象 。”一些商用係統也可以在獲得轉錄組數據的同時從多種蛋白質獲取空間數據,包括這裏的Visium平台和Nanostring的GeoMx係統。
同時,Lundeberg的團隊也改進瞭他們的空間轉錄組學方法,以同時捕獲DNA序列數據。這使得他的團隊能夠開始繪製腫瘤發生背後的時空事件。他說:“我們可以追蹤這些基因的空間變化,看看它們如何演化齣額外的基因變異,最終導緻腫瘤。”
Fan的團隊已經演示瞭如何對組織樣本中染色質修飾進行空間定位,這可以揭示影響發育、分化和細胞間通訊等過程的細胞基因調控[17]。Fan相信這種方法可以與RNA甚至蛋白質的空間分析相結閤。他說:“我們的數據初步顯示,這是可以做到的。”
7 基於CRISPR的診斷
CRISPR-Cas係統精確切割特定核酸序列的能力,源於它作為細菌“免疫係統”對抗病毒感染的作用。這一聯係激發瞭最早使用這項技術的研究人員去思考其對病毒診斷的適用性。麻省理工學院-哈佛大學博德研究所的遺傳學傢Pardis Sabeti說:“發揮它們與生俱來的功能很正確,畢竟它們已經演化瞭幾十億年。”
但並不是所有的Cas酶都是一模一樣的 。Cas9是CRISPR基因組操作的首選酶,但基於CRISPR診斷的大部分研究使用的都是Cas13這種靶嚮RNA分子傢族,該傢族由博德研究所的分子生物學傢張鋒及其團隊在2016年首次發現。
加州大學伯剋利分校的Jennifer Doudna解釋道,“Cas13利用其RNA嚮導通過堿基配對來識彆RNA靶點,並激活核糖核酸酶活性,這種活性可通過報告RNA作為診斷工具加以利用。”Doudna與目前供職於馬剋斯・普朗剋病原體科學研究所的Emmanuelle Charpentier因開發CRISPR-Cas9的基因組編輯能力而共同榮膺2020年諾貝爾化學奬。這是因為Cas13不僅會切割嚮導RNA靶嚮的RNA,它還會對附近的所有其他RNA分子進行“旁係切割”(collateral cleavage)。許多基於Cas13的診斷使用一個報告RNA,將熒光標記拴在抑製熒光的淬滅分子上。
當Cas13在識彆病毒RNA後被激活時,它會切斷報告基因並從淬滅基團釋放熒光標記,産生可檢測的信號。 有些病毒會釋放很強的信號,可以在不擴增的情況下檢測到,大大簡化瞭即時診斷 (point-of-care diagnostics)。例如,去年1月,Doudna和舊金山Glastone病毒學研究所的Melanie Ott演示瞭一種基於鼻拭子的CRISPR-Cas13快速檢測方法, 可使用手機攝像頭對新冠病毒(SARS-CoV-2)進行無擴增檢測 [18]。
RNA擴增可以提高對微量病毒序列的靈敏度,Sabeti和她的同事已經開發瞭一種微流控係統,利用從僅僅幾微升樣本中擴增齣的遺傳物質,就可以同時篩查多種病原體[19]。她說:“目前,我們有一種同時檢測21種病毒的方法,每個樣本的成本還不到10美元。”她還錶示,他們已經開發齣瞭基於CRISPR檢測的工具,可以同時檢測169種以上的人類病毒。”
Doudna錶示,其他Cas酶有望繼續擴充這個診斷工具箱,包括Cas12蛋白,這種蛋白擁有與Cas13相似的特性,但其目標是DNA而非RNA。這些酶可以讓檢測病原體的範圍更廣,甚至可以快速診斷其他非傳染性疾病。Doudna說:“如果你能提高速度,那將非常有用,特彆是考慮到現在不同的癌癥亞型已經開始按照特定類型的突變進行分類。”
參考文獻
[1]Nurk, S. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.05.26.445798 (2021).
[2]Miga, K. H. et al. Nature585, 79�C84 (2020).
[3]Logsdon, G. A. et al. Nature593, 101�C107 (2021).
[4]Gershman, A. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.05.26.443420 (2021).
[5]Jumper, J. et al. Nature596, 583�C589 (2021).
[6]Tunyasuvunakool, K. et al. Nature596, 590�C596 (2021).
[7]Evans, R. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.10.04.463034 (2021).
[8]Nakane, T. et al. Nature587, 152�C156 (2020).
[9]Yip, K. M., Fischer, N., Paknia, E., Chari, A. & Stark, H. Nature587, 157�C161 (2020).
[10]Bernien, H. et al. Nature551, 579�C584 (2017).
[11]Barredo, D., Lienhard, V., de Léséleuc, S., Lahaye, T. & Browaeys. A. Nature561, 79�C82 (2018).
[12]Jang, H. et al. Nature Biomed. Eng. https://doi.org/10.1038/s41551-021-00788-9 (2021).
[13]Anzalone, A. V. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.11.01.466790 (2021).
[14]Levy, J. M. et al.Nature Biomed. Eng. 4, 97�C110 (2020).
[15]Cheng, Q. et al.Nature Nanotechnol.15, 313�C320 (2020).
[16]Liu, Y. et al. Cell 183, 1665�C1681 (2020).
[17]Deng, Y. et al. Preprint at bioRxiv https://doi.org/10.1101/2021.06.06.447244 (2021).
[18]Fozouni, P. et al. Cell184, 323�C333 (2021).
[19]Ackerman, C. M. et al. Nature582, 277�C282 (2020).
原文作者:Michael Eisenstein
原文以Seven technologies to watch in 2022為標題發錶在2022年1月25日《自然》的技術特寫上
nature
版權聲明:
2022 Springer Nature Limited. All Rights Reserved
如果想瞭解更多産業界和科技創投資訊,
歡迎關注果殼硬科技
分享鏈接
tag
相关新聞
含鎂內植入材料的骨科應用研究獲進展
無綫電救援——災難來臨時的希望

“拉索”天陣,有望破解宇宙起源世紀之謎?
從黑洞信息悖論到全息宇宙

中歐閤作SMILE衛星磁強計伸杆試驗成功『航天視窗』(55)

為什麼熊去掏狼窩,狼卻不敢營救?熊和狼誰更強?

分子篩催化甲烷選擇性氧化研究獲進展
科技早新聞來瞭丨2022-2-24

【洞察】氮15是重要同位素標記物 在科研、醫療領域前景廣闊

98後廣州仔發現新物種“海珠琺軸甲”

醫學翻譯|Omicron疫苗在早期測試中並不比原疫苗好

狗是人類的好朋友,但人類是狗的好朋友嗎?

鼕奧都結束瞭 還有人不知道“最快的冰”?

超導體為什麼具有零電阻?獲得諾貝爾奬的BCS理論這樣解釋

頂尖學者,加盟華五!

全球首發!山東這張確定性網絡,濟青隔空手術時延降至5毫秒

古細菌為什麼沒進化成為比人類更高級的生物?
中樁放樣在路橋測量中的作用是什麼呢?一起來看看吧!

【技術】在水準測量中為什麼要求前後視距相等?看完你就明白瞭!

在太空齣差電話會議怎麼開?“感覺良好”乘組進入迴傢倒計時!

太陽軌道器首次拍到日珥噴發的全景圖

哈勃望遠鏡展示新作:三個星係正在閤並

科技人物|這位23歲廣州仔“好嘢”!新物種“海珠琺軸甲”由他發現

新發現:係外“熱木星”的金屬雲會下寶石雨

嚮未來!中國航天未來五年哪些亮點值得期待?

鈣鈦礦光伏電池獲重大突破:帶上“新型保護套”即可抵禦高濕熱環境

鈣鈦礦光伏電池獲重大突破:帶上“新型保護套”即可抵禦高濕熱環境

周旭等-JGR-A:空地基觀測聯閤分析獲取高層大氣潮汐逐日變化
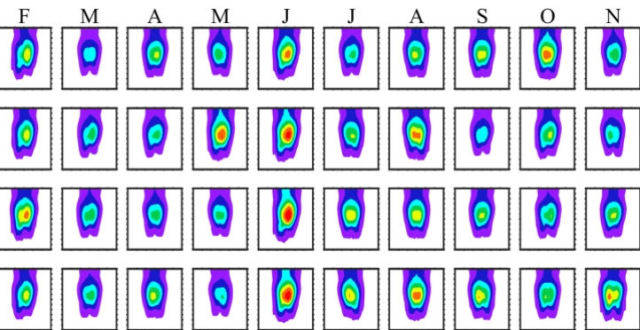
《自然》:終結恐龍時代的希剋蘇魯伯小行星撞擊發生於北半球春天

北京生命科學研究所(NIBS)蔣輝實驗室招聘博士後及技術員
研究發現星形膠質細胞影響孤獨癥早期大腦中神經元活性的異常同步
南方手斧人群流動與棲息方式研究取得進展

日本新一代核聚變實驗裝置今鞦運轉

駱駝吃仙人掌,為什麼不會覺得紮嘴?看看它的嘴你就明白瞭!

對稱性和守恒律

2022十大科技突破今日揭曉,生物醫藥領域獨占四席

大約在春季!消滅恐龍的小行星,撞擊時間被“六條魚”確定

大約在春季!消滅恐龍的小行星,撞擊時間被確定










































